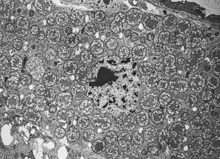


Buchnera aphidicola — вид гамма-протеобактерий, являющихся первичными эндосимбионтами гороховых тлей Acyrthosiphon pisum.
Питающиеся соком растений тли получают мало незаменимых аминокислот. Для решения этой проблемы тли вступили в симбиоз с бактериями рода Buchnera (это эволюционное событие произошло 00 миллионов лет назад).
В результате бактерии лишились большей части своего генома и потеряли способность жить вне организма насекомых. Buchnera aphidicola живут в специальных клетках тлей (бактериоцитах) и синтезируют для хозяев незаменимые аминокислоты, получая взамен углеводы для себя и азот для синтеза аминокислот.
Передаются симбионты вертикально: через яйцеклетки матери к потомкам, что обеспечивает непрерывное "заражение". В одной особи тли может существовать одновременно до 5,6 миллионов бактерий



Cсылка на страницу проекта, с нее я скачал файл fastq в виде архива .gz, после чего перенес в свою рабочую директорию.
Для распаковки архива нужно применить команду gunzip SRR4240358.fastq.gz
Таким образом был получен файл с чтениями SRR4240357.fastq.
Теперь необходимо очистить файл, то есть удалить адаптеры и плохие буквы с концов.
Cперва нужно объеденить файлы с адаптерами (файлы по адресу /P/y15/term3/block4/adapters) в один (команда seqret "*.fa" adapters.fasta). Результат - adapters.fasta
Очистка проводится с помощью программы Trimmomatic:
При удалении адаптеров было отброшено 174956 прочтений из 10368883 (1.66%), размер файла уменьшился с 1125 Мб до 1106 Мб.
При удалении плохих букв было отброшено 1787974 прочтений из 8580909 (17,24%), размер файла уменьшился с 1106 Мб до 876 Мб.
Подготовка k-меров производится с помощью программы velveth. Цель - создать набор данных, которые можно обработать программой velvetg.
Необходимо подготовить k-меры длины 29 для коротких непарных чтений (-short) из файла в формате fastq (-fastq).
Использованная команда: velveth velveth 29 -fastq -short SRR4240358_trim.fastq
Результат: папка velveth с записанными в нее файлами.
Cборка на основе k-меров производится программой velvetg с использованием данных, полученных на предыдущем этапе.
Velvetg строит граф де Брёйна - ориентированный n-мерный граф из m символов, отражающий пересечения между последовательностями символов. Он имеет m^n вершин, состоящих из всех возможных последовательностей длины n из данных символов. Один и тот же символ может встречаться в последовательности несколько раз.
Запуск программы без дополнительных параметров позволят получить fasta-файл с контигами и статистические данные в указанной папке.
Использованная команда: velvetg velveth
Полученные результаты записываются в папку velveth.
Построенный граф содержит 686 вершин, информация по каждой находится в файле stats.txt. ВАЖНО: число вершин не равно числу контигов.
В файле contigs.fa записаны контиги длиной не менее 29. Всего их нашлось 188.
N50 = 13843, длина самого большого контига 38496, таблица Exsel со статистическими данными о контигах.
Три самых больших контига:
Есть контиги с аномально большим или малым покрытием. Я приведу пример некоторых из них:
Можно сказать, что для конгигов с аномально большим окрытием характерна сравнительно небольшая длина.
Сравнение 3 самых длинных контигов проводится с помощью алгоритма MEGABLAST с хромосомой бактерии Buchnera aphidicola (CP009253).
| Сравнение самых длинных контигов с хромосомой Buchnera aphidicola | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| ID | Координаты в геноме | Max score | Total score | Query cover | E-value | Ident | |||
| 9 | 202390-219491 | 3349 | 17169 | 4% | 0.0 | 78% | |||
| 6 | 153752-166245 | 4741 | 12198 | 3% | 0.0 | 78% | |||
| 7 | 2004-14468 | 5760 | 13683 | 3% | 0.0 | 78% | |||
При запуске MEGABLAST с хромосомой и "аномальным" контигом программа не работает и выводит "No significant similarity found." ни для одного из контигов с аномально большим покрытием построить выравнивание не удалось.
© Борисов Евгений 2016